|
文獻解讀丨(CELL=66.85)通過凍融脂質體-蛋白質復合物輕松制備脂質體介導的蛋白質遞送系統
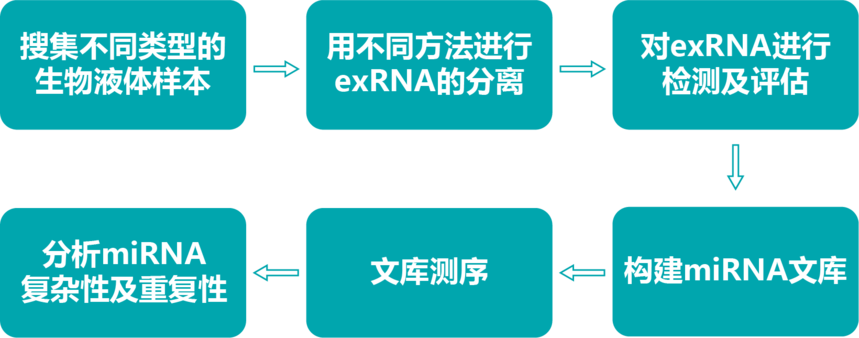
期刊:CELL 發表日期:2019 IF:66.85 摘要 由于對細胞外RNA (exRNA)分離方法的性能缺乏了解,導致細胞內部和跨研究的重復性差,阻礙了exRNA領域的進展。通過檢測評估細胞外囊泡(EV)、核糖核蛋白(RNP)和高密度脂蛋白(HDL)的特異性miRNA,確定每種分離方法對不同exRNA載體亞類的相對效率。  文獻思路: 1 對不同類型液體樣本進行exRNA分離,miRNA建庫測序后進行數據分析 2 不同種類RNA的分布因生物液體和exRNA分離方法而不同 3 qRT-PCR鑒定exRNA分離和定量的變異來源 4 RNA-seq分析顯示不同種類RNA的分布因生物液體、供體/細胞系exRNA分離方法而不同 5 small RNA-Seq中miRNA 的結果之一:miRNA的Complexity與miRNA的測序深度相關 6 利用miRNA的Complexity、表達量和重復性指標來評估exRNA分離方法的性能 (1)樣本 plasma 血漿、serum 血清、urine 尿液:來自10名男性或女性,在Lab5進行收集; Bile 膽汁:三名疑似患有膽管癌的捐贈者(P1–P3)和一個健康的成人捐贈者(P4); 細胞培養條件培養基(CCCM):在三個實驗室收集了三種細胞類型 human embryonic stem cells (hESCs, Lab5); primary neonatal rat ventricular myocytes (NRVMs, Lab2); human cholangiocarcinoma cells (KMBCs, Lab6)。 (2)exRNA分離細節 膽汁(200μL input):ExoRNeasy, miRNeasy, Exiqon, Ultra, and Millipore; 細胞培養條件培養基 (4 mL input):ExoRNeasy, Ultra, Millipore, and ExoQuick; 血漿和血清 (500μL input):ExoRNeasy, miRNeasy, Exiqon; 尿液 (500μL input):ExoQuick, ExoRNeasy, Homebrew, Millipore, miRNeasy, and Ultracentrifuge。 (3)small RNA文庫構建 所有實驗均在同一實驗室進行。 對于血漿、血清和CCCM,對來自一個實驗室的三個重復exRNA分離物和來自其他實驗室的一個重復進行了測序; 對于膽汁,分析了四個樣本,因為在qRT-PCR實驗后,其他兩個樣本材料不足;對于尿液,所有樣本來自合并后的女性和男性,一個女性樣本一個男性樣本。 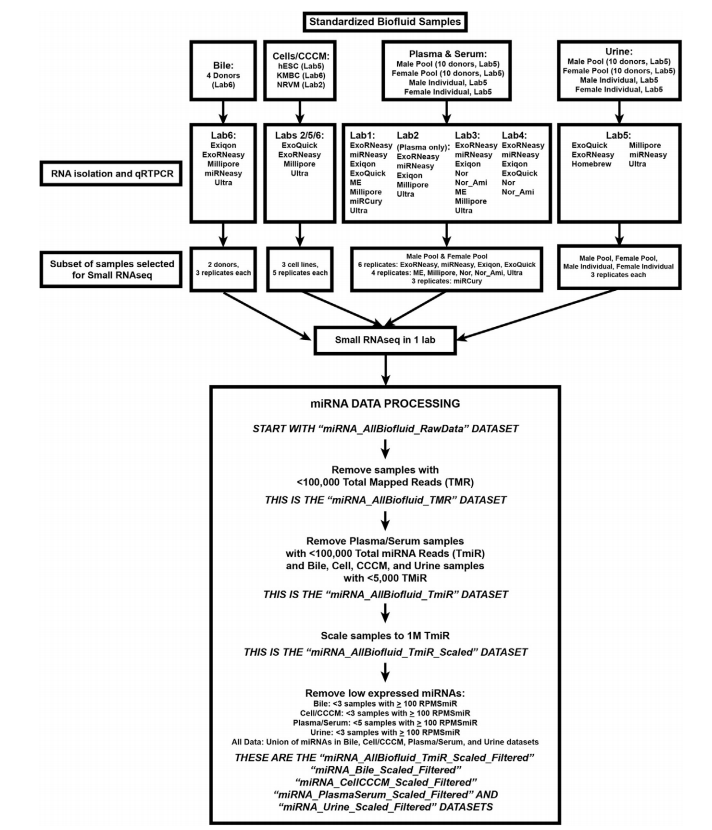  不同類型的生物液體樣本,用不同方法分離得到的exRNA片段分布和產量不同。 RNA大小分布差異表現為:所有血清和血漿樣本均表現為雙峰分布,在ExoQuick和ExoRNeasy樣本中較短(<200nt)占優勢,較長(>200nt)RNA在miRNeasy樣本中占優勢,這兩個峰在Exiqon、Nor和Nor_Ami樣本中都有很好的表現; Ultra的結果變化最大,實驗室2的樣本中RNA大多較短,實驗室5的短rna和全長18S和28Srna,實驗室6的總產量較差;尿液中,RNA大小分布相似,但不同方法的產量不同,ExoQuick、 Ultra和Millipore的產量低于ExoRNeasy、miRNeasy和Homebrew; 此外,使用RiboGreen (Thermo Fisher)測量exRNA濃度,但結果顯示通過生物分析儀、qRT-PCR和小RNA序列清晰檢測到的RNA,無法在RiboGreen檢測到。因此,研究者不建議使用RiboGreen來定量分離exRNA。 采用三個miRNA,這三個miRNA都曾經在多篇文獻中被報道存在于細胞分泌的外泌體中:let-7a-5p,miR-16-5p,miR-223-3p。qRT-PCR數據采用方差分析法進行分析,變異性使用the sum of squares divided by the total error。 檢測過程和結果為:膽汁樣本,exRNA分離在同一個實驗室中進行,差異的最大來源是樣本(P1-P4),其次是exRNA分離方法,三種miRNA之間的變異較低; 尿液樣本,exRNA分離在同一個實驗室中進行,變異的最大來源是分離方法,其次是生物流體供體的性別,目標miRNA和樣本的貢獻可以忽略不計; CCCM樣本,其中實驗室是變異性的最大來源,其次是靶miRNA、exRNA分離方法和源細胞系; 血清和血漿樣本:qRT-PCR數據同時采用兩種方法同時進行分析,變異性的最大來源是靶標miRNA,其次是實驗室和exRNA的分離方法。 這些結果表明:當檢測少量miRNAs的qRT-PCR數據時,exRNA分離方法是除CCCM外的所有生物液體變異的重要來源; 在所有的多實驗室實驗中,實驗室變量對變異性的貢獻顯著,使用相同的qRT-PCR機器的兩個實驗室產生了最相似的結果。這一觀察結果表明,qRT-PCR過程,而不是exRNA分離過程,可能是實驗室間實驗室變異的主要來源; 對于CCCM和血清/血漿,靶miRNA也是變異的一個重要來源,這表明檢測的三個miRNA可能攜帶在這些生物液體的不同成分中。 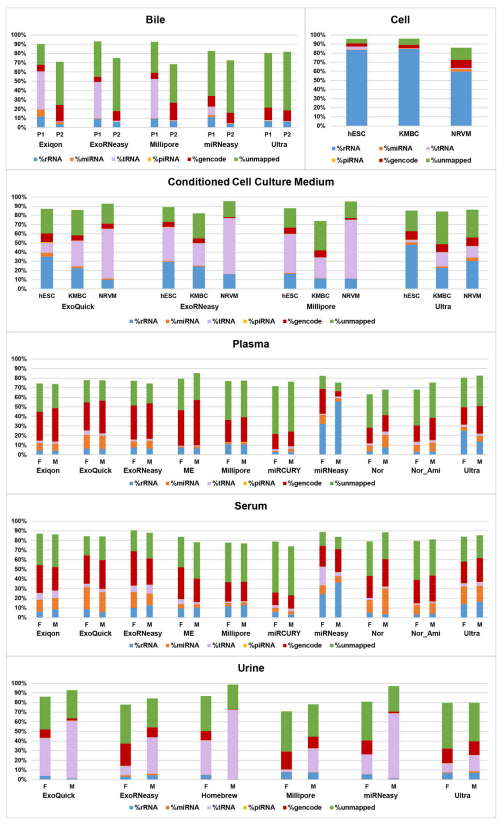 作者統計了不同RNA種類的比對率:rRNA, miRNA, tRNA, piRNA其它基因組序列, 以及未比對上的reads,比較了每種生物液體類型中不同方法中每種RNA種類的百分比。 對于膽汁,Exiqon的%miRNA顯著高于所有其他的方法,而P1的%tRNA顯著高于P2。RNA種類在所有細胞RNA樣本中的分布相似,以rRNA序列為優勢; 對于CCCM,Ultra的%tRNA顯著低于其他方法,而ExoQuick和Ultra的%miRNA顯著高于ExoRNeasy和Millipore; 對于血漿和血清,它們之間的RNA種類分布相似,miRNeasy和Ultra樣本的%rRNA顯著高于其他方法,ME、Millipore和miRCURY的%miRNA顯著低于Exiqon、ExoQuick、ExoRNeasy、miRNeasy,Nor, and Ultra; 對于尿液樣本,總體上有很高的%tRNA,其中ExoRNeasy和Ultra方法產生最高(但仍然相當低)的%miRNA。 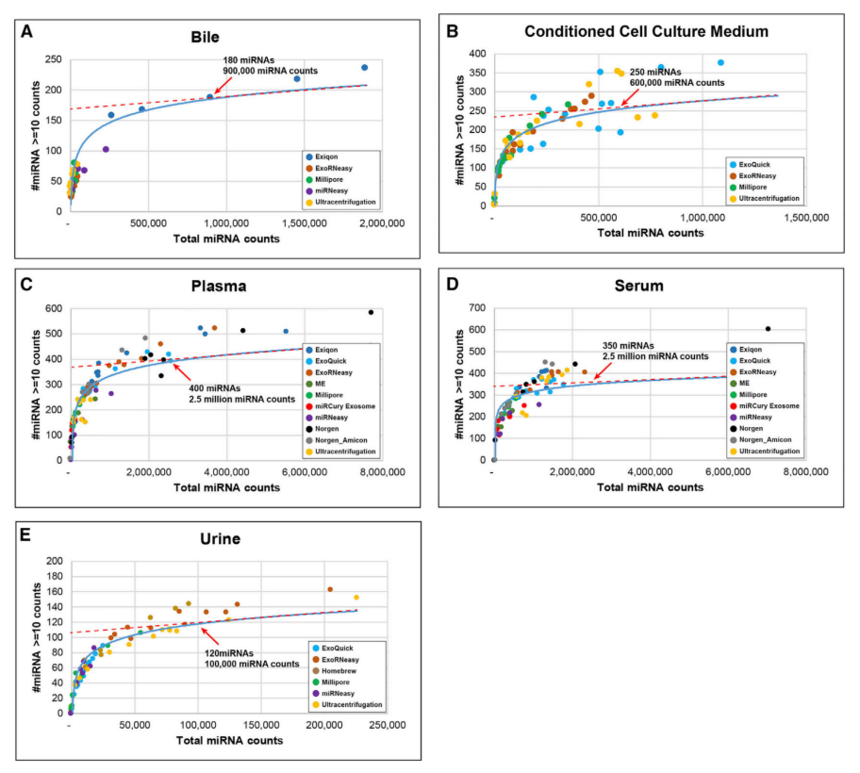 作者對small RNA-Seq 中miRNA進行分析發現,miRNA的Complexity與miRNA的測序深度相關。Complexity是指number of miRNAs with >= 10 raw counts。miRNA的Complexity與測序深度(total miRNA read count)正相關。 對于每種生物液體,Complexity會逐漸穩定在一個特定的miRNA測序深度,也即測序飽和度。不同的液體類型,飽和度不一樣。 對于膽汁樣本,180 miRNAs at 0.9 million total miRNA counts,對于膽汁,只有Exiqon庫達到了這個飽和; 對于CCCM樣本,250 miRNAs at 0.6 million total miRNA counts;對于血漿樣本,400 miRNAs at 2.5 million total miRNA counts; 對于血清樣本,350 miRNAs at 2.5 million total miRNA counts;對于尿液樣本120 miRNAs at 0.1 million total miRNA count。 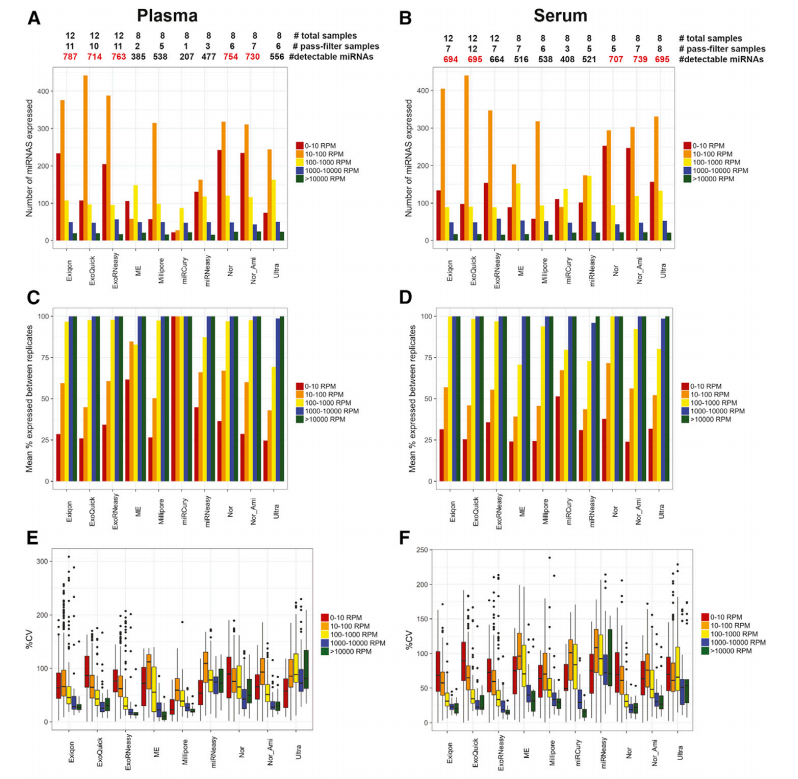
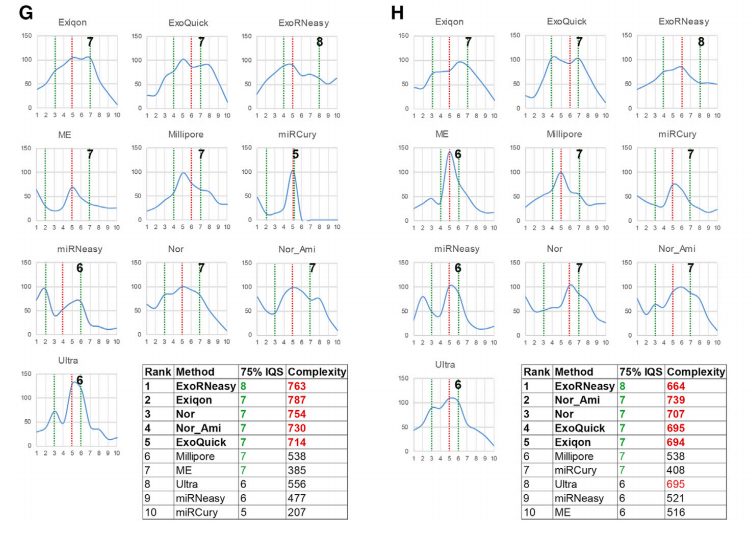
mean number of detected miRNAs; mean percentage of replicates in which a given miRNA was detected; percent coeffificient of variance (%CV) across replicates; a combined expression reproducibility score, which we term the integrated quality score (IQS)。 由于檢測到的miRNA的數量及其在技術重復中一致表達的概率取決于平均表達水平,因此根據每種生物液體和分離方法的平均miRNA表達水平(以每百萬[RPM]miRNA計數表示)展示結果。 對于每種生物液體及分離方法,每個miRNA的IQS是exRNA分離試劑盒的平均表達分位數和%CV分位數的和。在所有對給定miRNA有可測量表達的分離方法中,平均表達分位數和%CV分位數的評分范圍為1-5:1為最低平均表達或最高%CV,5為最高平均表達或最低%CV。計算在至少四種方法中具有可測量表達的miRNA的IQS值,因此,對于給定的分離方法,更高的IQS表明更高的表達和更高的重現性,具有更好的分析性能。 通過比較不同exRNA分離方法中IQS值的分布,作者可以很容易地選擇最佳的方法來分離目標miRNA列表或為所有miRNA。理想的分離方法上圖(G和H)應該有明顯的左偏斜,表明具有更高表達和重復性的miRNA比例更高。計算第75百分位IQS(IQS75)作為左偏度的指標,并結合每種分離方法的complexity,可以對每種被測試的exRNA方法的性能進行排序。使用這些指標,血漿和血清的檢測結果相似,與其他方法相比,ME、Millipore、miRCURY、miRNeasy和Ultra與其他方法相比顯示出明顯較低的復雜性。除血漿中的miRCURY試劑盒外,在重復中表達的miRNAs的百分比與平均表達水平呈正相關 結論 這項研究系統性地比較了不同類型的生物液體樣本通過不同的分離方法,exRNA的miRNA-seq不同: 測序的復雜性和可重復性因分離方法而異; 不同分離方法對exRNA載體的獲取不同; exRNA分離方法的性能因生物液體和RNA的種類而不同; miRDaR能夠定制選擇最佳的exRNA分離方法。 從而為exRNA的分析研究中,提高檢測的準確性和重復性而選擇定制“最佳分離exRNA方案”提供了指導。 |